 醫學研究部(Department
of Medical Research)
醫學研究部(Department
of Medical Research)共 同 研 究 室 電 子 報
第九十四期 OCT.10.2021
本期目錄
下期主題
快速連結
近年來,流式細胞儀的發展已達可同時分析數十個螢光,如何客觀自動化地分析這些高參數數據是如今分析軟體計算技術開發的重點。本期電子報將介紹多種流式細胞高參數數據的分析,協助大家探索未知的領域,增加研究的質與量。流式細胞核心目前設有FlowJo分析軟體可供使用,亦提供流式細胞實驗數據委託分析之服務,歡迎有需要之同仁與我們聯絡。下一期電子報主題為「ImageXpress NANO高內涵影像系統最新研究應用」,敬請期待,並竭誠歡迎您訂閱共同研究室電子報以收取儀器介紹、研究新知、與每月訓練課程資訊,更歡迎您與我們聯絡,給予我們建議與鼓勵。
另基因定序暨生化核心將於10/15日開放外泌體(Extracellular
vesicles )萃取服務。外泌體為近期熱門研究話題,研究領域廣泛,但由於外泌體的多樣性來源(如:血液、培養液、尿液),如何純化外泌體為首當其衝的課題。本核心提供四種外泌體純化方法,同仁可根據樣本特性和實驗需求選擇最佳純化方式,相關資訊請於核心官網查詢。預約服務請填寫Redcap表單,有相關問題請洽技術員(分機63271
薛先生)。希望我們所提供的訊息對您的研究有所助益,服務品質也令您滿意,為了共研長期的經營運作,請您於發表文章時惠予致謝共同研究室,作為服務成效評鑑之用。
TOP
必帝(BD)股份有限公司 胡渝翔專員
在過去的二十年間,流式細胞儀技術有著突飛猛進的發展,從早期僅可以同時偵測四個螢光,到現今最多可同時偵測至數十個螢光,流式細胞儀一次測量更多參數的能力不斷增強,許多高參數數據每天都不斷地產生,這些數據無法再使用傳統的(主要是手動的)方式進行完整深入的分析。因此新穎的計算技術不斷被開發出,讓研究人員可以一種自動化且客觀的方式分析,透過將資料視覺化 (Visualization)的處理,幫助解讀高參數的細胞數據。
在過去我們根據免疫學的知識與透過肉眼觀察將細胞分群,並對數據進行圈選分析,但這樣的方式限制了我們的分析,並且導致主觀分析與偏見。用手動分析流式細胞數據時,研究人員通常只會查看自己喜歡的細胞類型,而這樣做會從分析中丟棄許多其他細胞。另外,在圈選步驟中,細胞標記會根據實驗假設被歸類,且通常在之後的圈選階段不再去重新考慮這些標記的分布情況。同時,主觀性也發生在選擇參數組合階層順序的級別,以及在分析中指定的每個圈選的形狀和邊界上。
透過計算生物學的方式進行流式細胞高參數分析能變得客觀許多,並且不因任何已知的細胞類型而產生分析偏差。演算法計算數據本身,並評估樣品中所有細胞的相似性,也同時考慮了所有標記,這使得我們能發現新的細胞族群或生物標記物,而這些新的細胞族群或生物標記物通常在手動分析中被輕易丟棄。
在此我們簡介廣為研究使用的 tSNE 降維演算法在FlowJo 中進行流式細胞高參數數據的流程,包含以下從Cleanup > Concatenate >Dimensionally-reduce > Cluster >Gate > Comparisons> 這幾個主要分析階段以及注意事項。
T-分佈式隨機鄰居嵌入(tSNE)是用於降維的演算法,允許在較少的維度下可視化複雜的多維數據,同時仍然保持數據的結構。 tSNE 是一種無監督的非線性維度減少算法,可用於在降低維度的數據空間中視覺化高維度的流式細胞數據。 tSNE從用戶定義的流式細胞參數中選擇生成兩個新的參數創造出高參數數據的二維視圖,讓我們可以識別出有興趣的或是罕見的細胞族群,降低傳統2維分析需要數張圖形才能表達的概念。tSNE是一種功能強大的可視化技術,但對運算的輸入數據非常敏感。一個粗糙不嚴謹的數據將會產出糟糕的輸出結果,若不經過標準化的儀器設定與QC以及標準化實驗檢體染色製備的操作流程與產出檔案的整理,相同的實驗但不同的組別在分析上可能產出非常不理想的結果。由於篇幅有限,我們謹在此介紹實驗做完後產出的FCS檔在軟體上的處理。
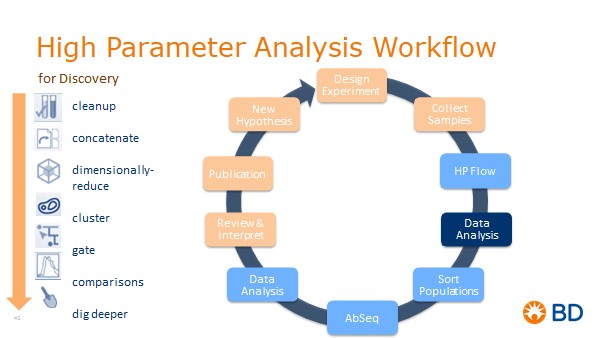
圖一:高參數分析流程圖
進行tSNE 降維演算法分為Cleanup > Concatenate >Dimensionally-reduce > Cluster >Gate > Comparisons 這幾個主要分析階段(圖一)。
-
Cleanup:主要是將 doublets,死細胞與碎片等干擾去除,不去影響到資料運算。可以提高tSNE算法輸出的品質。 此外,最好僅包括感興趣的細胞(例如,如果T細胞是主要的目標,就只圈選CD3 +的細胞進行演算)。
-
Concatenate:將所有待分析的樣本資料合併到單個文件中以進行運算,在此階段通常需要先使用DownSample工具,將分析樣本細胞數進行正常化讓各個實驗組別有相同的細胞數量與分布,使結果不因各實驗組別細胞數不同而失真。
-
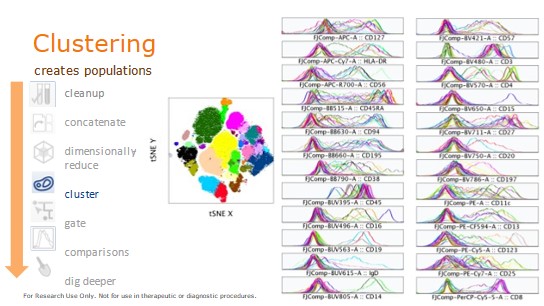
Dimensionally-reduce(降維):利用tSNE演算法,在高維度中具有類似參數的細胞群在降維後的二維數據空間內可以被清楚的以島的形狀顯示出(圖二)。

圖二:利用tSNE演算法進行降維
4.Cluster:使用演算法進行自動聚類,常見的聚類演算法如(FlowSOM,PhenoGraph,X-shift利用不同的模型進行聚類)。自動細胞群體聚類的一個重要方面在於選擇聚集數量。有些聚類工具將聚集數量明確作為輸入(例如FlowSOM)。其他工具則透過與聚集數量直接相關的參數(例如,基於密度的聚類算法中的鄰域大小)作為計算。大部分聚類算法屬於無監督的機器學習算法,這意味著沒有對任何細胞進行標籤或分組。只有根據流式細胞儀得到的測量結果和一些參數設置(例如,預期的聚集數量)作為輸入進行演算輸出。因此,大多數聚類演算法,只識別具有類似標記表達的細胞群,並未將這些細胞群進行標記,研究人員仍需要確認細胞標記去識別對應到已知的細胞群。
5.Gate:圈選疊圖,將cluster後的不同分群與tSNE疊圖可得到各個不同的cluster 在tSNE上的部份情況,若是再與傳統的手動圈選進行疊圖可協助辨識cluster為何種細胞群(圖三)。
圖三:Cluster後的族群與tSNE疊圖顯示各細胞群在島圖的分佈
6.Comparison:Cluster後的目標細胞群可藉由在concatenate中將各組不同的實驗結果合併圈選分析出。藉由圈選分析合併前的組別可以比較在各個組別中不同細胞群的細胞標記表現量的差異從而找出目標細胞群的變化,甚至可以發現以前未發現的細胞群進行跟深入的探索(圖四)。
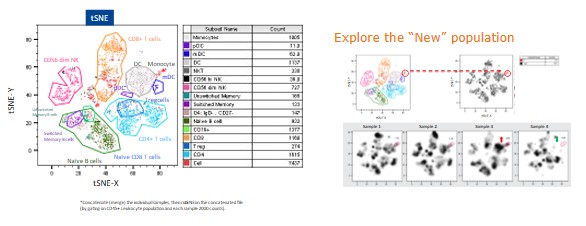
圖四:將各組不同的實驗結果以tSNE合併分析
最新研究趨勢將流式分析結果與演算法結合,依照每顆細胞所展現的螢光特性進行計算出視覺化的空間分布,幫助了解樣本中所有細胞分群,擴大觀測視野。期望透過良好的分析工具,能支持科學發展,並協助大家探索未知的領域,增加研究的質與量。
儀器訓練課程:課程網路報名
|
10月27日 |
ImageXpress NANO高內涵影像系統儀器訓練 |
研究新知課程:課程網路報名
|
10月28日 |
Targeting neoantigens for precision immune cell therapy against cancers |
歡迎您訂閱共同研究室電子報以收取儀器訓練與研究新知課程講習相關資訊 。
為持續提供優質之研究服務,便於日後聘用專職技術人員、購置新儀器、現有儀器汰舊換新與維護保養等等,敬請於使用共同研究室資源並發表論文時,於論文致謝(Acknowledgement)處加入致謝共同研究室之文句,並於論文發表時通知共同研究室管理人員。致謝文句請依實際使用情形書寫,或請參考以下範例:We
thank the staff of the Core Labs, Department of
Medical Research, National
Taiwan University Hospital for
technical support.