 醫學研究部(Department
of Medical Research)
醫學研究部(Department
of Medical Research)共 同 研 究 室 電 子 報
第六十八期 AUG.10.2019
本期目錄
下期主題
快速連結
開放資料是為了促進學術研究的透明度與研究成果的再現性,而累積的資料,也推動了以資料為導向的研究方式。在本期內容,說明了開放資料所能帶來的好處,及如何去探索這些科學資料,以利進一步的再利用並發現新的研究成果。下一期電子報主題為「掠奪性期刊案例分享」,敬請期待,並竭誠歡迎您訂閱共同研究室電子報以收取儀器介紹、研究新知、與每月訓練課程資訊,更歡迎您與我們聯絡,給予我們建議與鼓勵。
TOP
醫學研究部 許家郎助理研究員
一項科學實驗的進行,常受一些不起眼的因素影響,例如操作的人員、儀器的品牌、甚至氣溫濕度等,皆有可能干擾實驗的結果,這些實驗的「過程」與「細節」,宛如一個黑盒子,對於這些因素的疏忽,往往影響到實驗結果是否能夠再度呈現。隨著科學資料造假與研究誠信的新聞,不斷上演,科學研究的「透明度」與「再現性」,引起大家的關注,並思考如何確保研究發表的品質。其中,高通量實驗方法,包含微陣列技術(microarray)、次世代定序與質譜分析等,已漸漸成為各類生醫研究的利器,除了許多因素會影響數據結果外,例如批次效應(batch effect)外,高通量實驗數據,需搭配後續的資料分析或統計方法來確保數據品質,才能獲取有用資訊。然而,不同的分析方法或參數的使用,將影響實驗結論,因此高通量實驗與數據分析的透明度與再現性,特別受到重視。
為了提高高通量實驗與數據分析的透明度與再現性,其中「開放資料 (open data)」是一項可以提高透明度的策略。簡單來說,就是將產出的資料公開與其他人共享。目前,幾個主要的出版社,如果實驗有牽涉到高通量實驗數據時,投稿時會要求須把原始數據公開,供他人檢視。由於資料的公開,各方人馬就可以針對數據重新檢視,探討文章中所使用的分析方法是否恰當。的確,面對「巨量」的資料時,最擔心的莫過於“Garbage in, garbage out”,如果沒有小心的檢視資料,或使用不適當的分析方法時,很容易得到錯誤的結論。過去有一篇發表在頂級期刊的研究,他們分析了許多RNA-seq資料並得到很亮眼的結論。但不久後,有人重新分析同批資料,發現當初資料並未處理好,經過重新處理後,當初所得到的結論根本只是批次效應所造成。另一個近期的例子:有一篇2018年發表在Nature雜誌,利用深度學習(deep learning)來預測餘震的研究[註1],拜在原始資料與分析編碼皆公開,因此被其他學者發現,研究設計上有數據洩漏(data leakage)的疑慮,造成預測效果特別佳,引來許多討論[註2]。由這幾個例子可知,藉由資料的公開與共享,的確可以提高研究的透明度。
開放資料除了提供檢視研究內容的機會外,所累積下來的龐大資料,也帶來其他的應用面,特別是以資料驅動(data-driven)研究方法為主的生物資訊與系統生物學領域。其實資料驅動的研究方法並非新穎的想法,只是在過去,資料來源主要是經由實驗所產出,或者特地蒐集來,過程費時且費工。但現在這資料爆炸的時代,加上開放資料觀念的驅使,資料取得並得更加容易,創造了更多想像與可能。因此,當心中萌生新的研究想法時,別急著動手做實驗,先找找是否有適當的資料,並經由資料分析來測試假設是否可行;或者收集相關的資料,並將資料整合分析,尋找新的研究標的。這樣的研究策略,確實可以節省不少時間與金錢,前提是需要知道去哪裡找資料以及一位好的生物資訊學/計算生物學家。
資料開放或共享的方式,可藉由存放在個人或實驗室網頁上,或者放置公開的資料庫。目前,如果研究成果牽涉到使用基因體(例如whole-genome sequence (WGS), whole-exome sequence (WES), SNP array, array CGH等)、轉錄體(例如RNA-seq, cDNA microarray等)、蛋白體或其他高通量實驗數據(例如ChIP-seq等)時,期刊通常會要求將原始數據放置公開資料庫,並取得一組獨立序號,才會被接受刊登或進一步的審查工作。有些人也許會擔心,資料尚未發表,資料公開是否會被他人搶先發表。關於這點,大部分的公開資料庫,皆有公開時間的設定,因此當成果尚未發表時,資料將會被設定成隱藏。
資料庫方面,如果先不討論蛋白質體資料,主要有兩大體系,一個是由美國NIH的NCBI(National Center for Biotechnology Information)所管理的系統,另一個則由歐洲EMBL(European Molecular Biology Laboratory)下的EBI (European Bioinformatics Institute)所管理的系統。如果依照資料特性,可以分成管制(access control)與非管制資料。管制資料通常是個人可辨識的基因型與表現型資料,例如WGS, WES和SNP array等,其餘的資料型態則稱為非管制資料。管制型資料,主要存放在NCBI下的dbGAP(The database of Genotypes and Phenotypes)與EBI下的EGA(European Genome-Phenome Archive)兩大資料庫中。由於資料是受到管制,資料取得需經嚴格審查,這過程類似倫理委員會的審查機制,確保資料合乎倫理規範 (圖1),因此需提出研究計畫書,描述研究目的與如何使用請求的資料等。經審查通過後,即可存取資料。而非管制資料,取得則相對容易,通常經查詢瀏覽後,即可直接下載。針對非管制資料,主要有兩個資料庫儲存此類型資料,分別是NCBI下的GEO (Gene Expression Omnibus) 與EBI下的ArrayExpress。由名子來看,可知當初主要是針對微陣列資料的存取,但隨著不同技術的發展,現在各類型的高通量資料皆可接受。雖是兩個獨立資料庫,但他們已通過資料交換協議,因此不論當初資料是從GEO或ArrayExpress上傳,在兩個資料庫皆查詢的到。另外,GEO上也提供了簡單分析工具,稱作GEO2R,可執行表現差異分析與查詢單基因在不同條件下的表現量(圖2),對於無任何資料分析基礎的人,可經此線上工具做初步分析。關於蛋白質體資料存放,主要是由ProteomeXchange協會制定規範,並可透過合作夥伴系統進行資料上傳,例如EBI的PRIDE或日本的JPOST(圖3)。資料與資源很多,因此可以多加利用這些資料庫進行探索,將「老」資料重「新」再利用。
註記:
[1]https://www.nature.com/articles/s41586-018-0438-y
[2]https://www.theregister.co.uk/2019/07/03/nature_study_earthquakes/
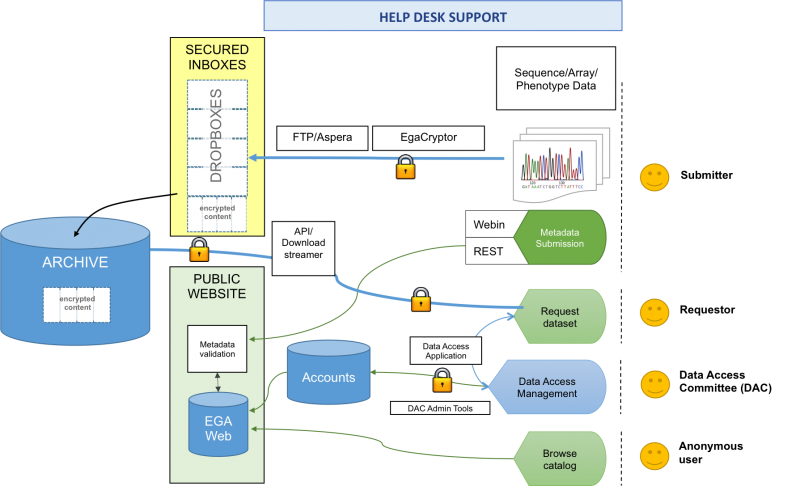
圖1. EGA系統概述。(來源: https://www.ebi.ac.uk/ega/)
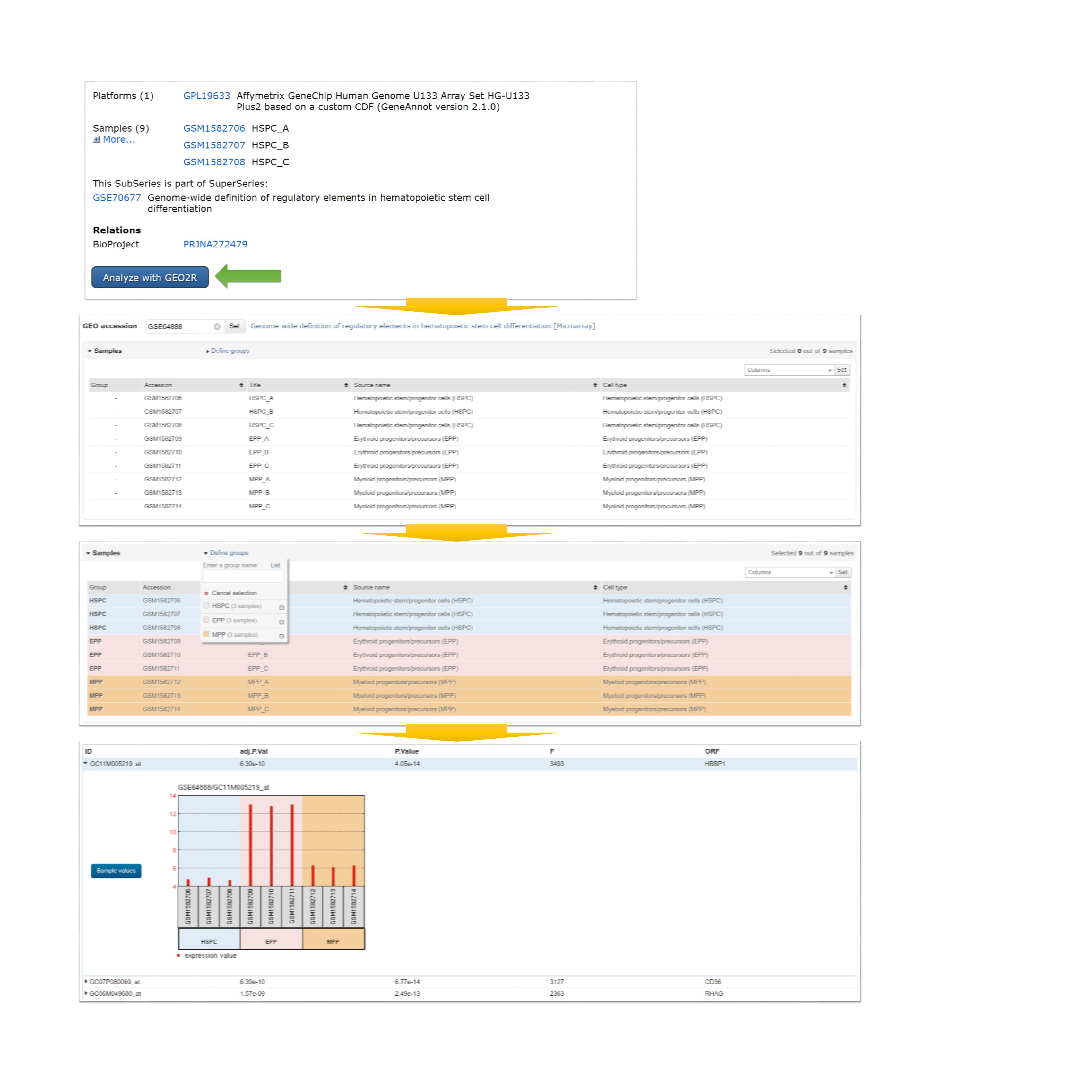
圖2. GEO上所提供的簡易的資料分析工具。
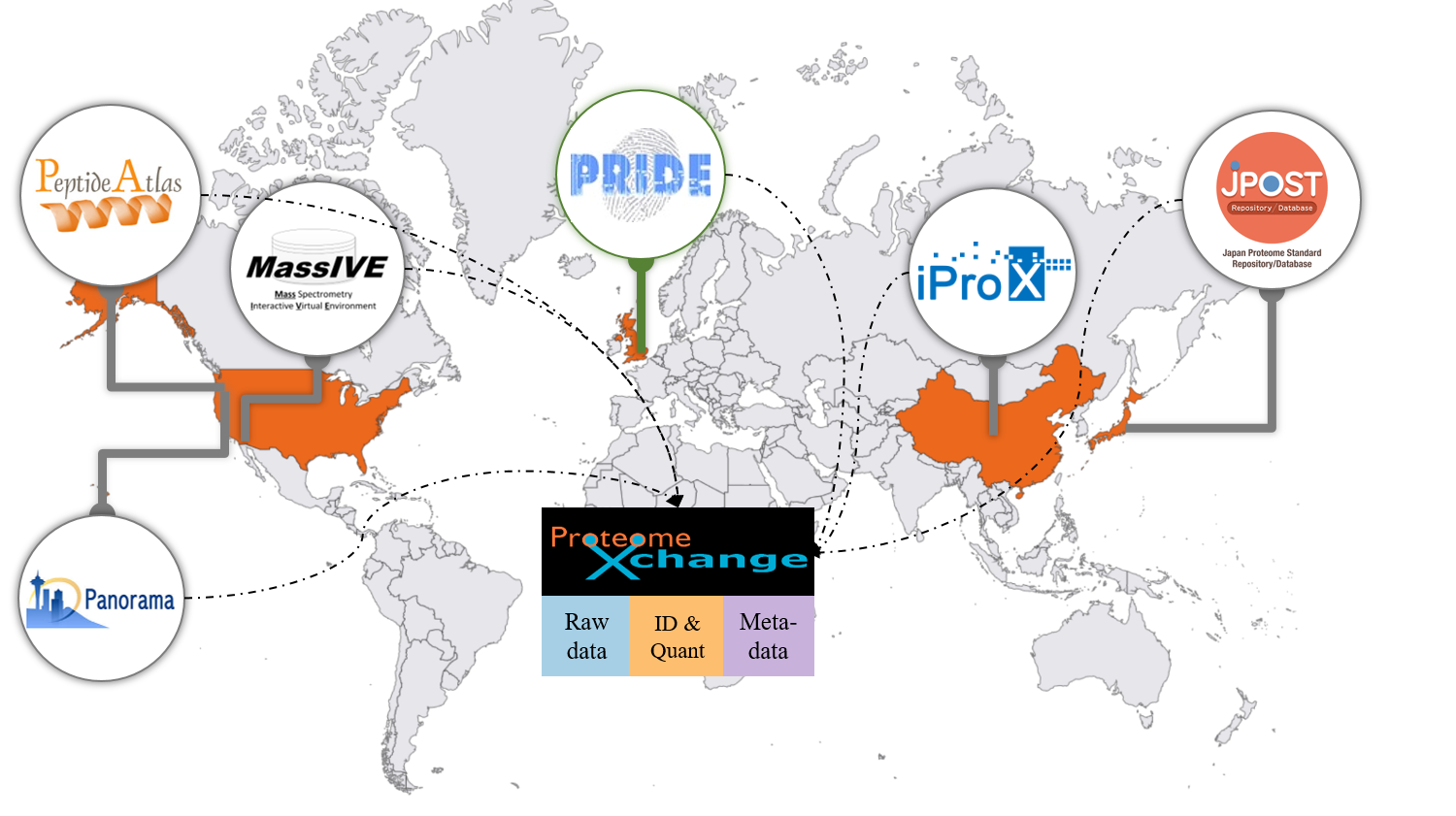
圖3. ProteomeXchange協會與夥伴機構。(來源: http://www.proteomexchange.org/)
儀器新知課程:課程網路報名
| 08月14日 | 同步定量PCR之原理與實機操作(AB QuantStudio 7 Flex Real-time PCR) | |
| 08月20日 | 新世代流式細胞儀 : BD FACSVerse系統原理介紹與應用CytoFLEX |
儀器新知課程:課程網路報名
| 08月13日 | 高通量資料與生物資訊分析服務說明會 |
歡迎您訂閱共同研究室電子報以收取儀器訓練與研究新知課程講習相關資訊 。
為持續提供優質之研究服務,便於日後聘用專職技術人員、購置新儀器、現有儀器汰舊換新與維護保養等等,敬請於使用共同研究室資源並發表論文時,於論文致謝(Acknowledgement)處加入致謝共同研究室之文句,並於論文發表時通知共同研究室管理人員。致謝文句請依實際使用情形書寫,或請參考以下範例:We
thank the staff of the Core Labs, Department of Medical Research, National
Taiwan University Hospital for technical support.